今回は、「実験解析のニュートレンド」の続編です。
前回は、「実験解析のニュートレンド」として、「シングルセルRNA-seq」についてお伝えしましたが、今回は、「シングルセルRNA-seq」について、続きをお届けします。
<3>シングルセルRNA-seq
1細胞ごとに別々に、RNA-seq法を用いて、トランスクリプトーム解析を実施するのが、シングルセルRNA-seqです。最初は、人の手で、顕微鏡を見ながら細胞を1つずつピッキングし、96Wellプレートの各Wellに1細胞ずつ入れて、解析していたそうです。気が遠くなりますね。その後、多くの細胞について実施できる装置が開発されたそうです。
シングルセルRNA-seqの方法はいくつかありますが、そのうちの1つとして、”Drop-seq”法について、その処理ステップをご紹介します(私のお勉強の成果でございます)。
(1)多くの細胞が集まった組織等を、1細胞ごとにバラバラにする
(2)特殊なマイクロ流路と、ゲルビーズを用意する。
ゲルビーズの表面には、ゲルビーズごとに異なるUMI(Unique Molecular Identifier)でラベルされたオリゴDNAプライマーが大量にくっついている(ゲルビーズの表面にオリゴDNAの毛が大量に生えている様なイメージ)。
(3)細胞を水溶液に浸し、ゲルビーズと細胞を、マイクロ流路に通し、1つの細胞に1つのゲルビーズをくっつける
(4)1細胞と1ビーズのペアを包む微小な水溶液の液滴(Droplet)を、オイルが入ったマイクロチューブにDropする。
オイルが入ったチューブには、マイクロ流路を通ってできた液滴が、次々とDropする。
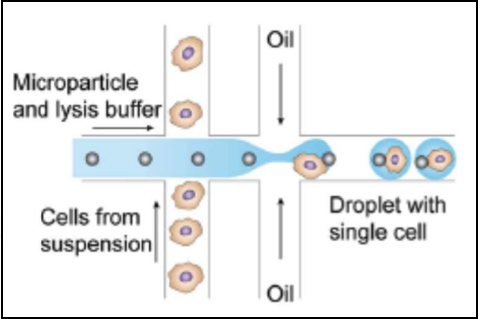
(5)液滴内で、細胞膜が溶け、UMIが付いたcDNAが合成される。
(6)PCR法によってcDNAを増幅する。
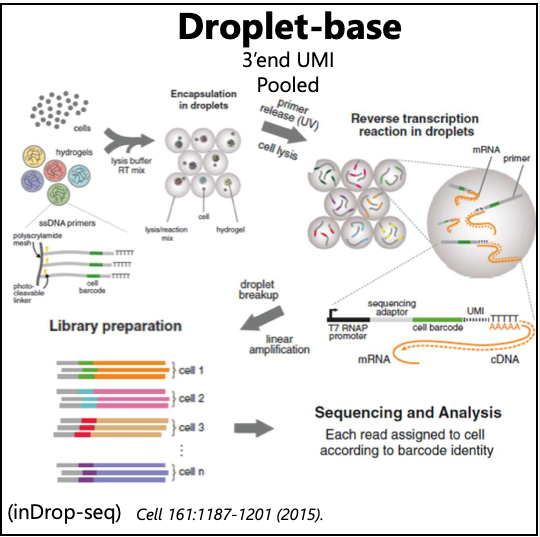
(7)次世代シーケンサーに掛ける
(8)シーケンサーの出力データを、UMIごとに分類する。1つのUMIは1細胞に対応している。
(9)メーカー提供のソフトや、サードパーティの「R」等を使って、mRNAの発現特徴について1細胞ごとにプロットしたグラフを作成する。
私の疑問点:(5)で、細胞膜を融解する方法、逆転写酵素を加えるタイミング
ひょっとしたら、これらはあらかじめゲルビーズに仕込んでおくか、あるいは水溶液に混ぜておくか、するのかもしれませんが、詳細不明です。
用語説明
・オリゴDNA: 短いDNA。(他の例:「オリゴ糖」は、短い糖)。
・プライマー: DNA合成処理の最初のきっかけを作るためのRNAもしくはDNA。
この”Drop-seq”法で特筆すべきなのは、
(1)ゲルビーズに、ビーズごとに異なるUMI(分子バーコード)でラベリングされたオリゴDNAプライマーを付けたこと。
(2)細胞1つに1つのゲルビーズを付けて、液滴に包み、液滴内で、細胞内のmRNAからcDNAを合成するという方法であること。
この方法は、従来の、マイクロプレートのWellを液滴に置き換え、液滴内で、細胞内のmRNAからcDNAを合成するという、とても斬新な方法であり、私はとても感銘を受けました。
この方法は、10X Genomics社の”Chromium”と、Bio-Rad社の”ddSEQ SCI”で、実現されています。
今回のテーマは、劇ムズでございました。今後はもっと敷居の低い話題にしたいと思います(心の声)。
私の感想としては、次世代シーケンサーの登場以降、研究者たちは、コンピュータによるデータ解析というお仕事が多くなったのでは? と思いました。コンピュータソフトの業界は目まぐるしく変化しますので、今後は、コンピュータ技術者が、研究者たちを支える仕組みが必要なのでは、と、感じました。また、コンピュータソフトでは、今、GitHub(ギットハブ)を使ったコラボレーションが多くなっていますので、生命科学でもそういったことが今後、多くなってくるかもしれませんね。
また、今勉強中の若い研究者(の卵)達は、今後、コンピュータの活用について、ますます重要性が増してくると思います。コンピュータの活用は、当たり前になってくると思います。コンピュータ技術者に頼るのではなく、ある程度のことは自分でできる様になる必要があると思います。頑張ってきただきたく思います。
参考:「やってみよう! single cell RNA-seq解析ことはじめ」
https://jp.illumina.com/content/dam/illumina-marketing/apac/japan/documents/pdf/webinar/professional-webinar-single-cell-190131.pdf

コメント